| Saúde |
Grupo da Unifesp investiga a diversidade de bactérias E. coli em pacientes hospitalizados
O intestino do ser humano é um ambiente repleto de microrganismos coletivamente chamados de microbiota intestinal. Essa flora, na maioria dos indivíduos, é benéfica à saúde, fortalecendo o sistema imunológico, melhorando o metabolismo e desempenhando diversas funções no organismo.
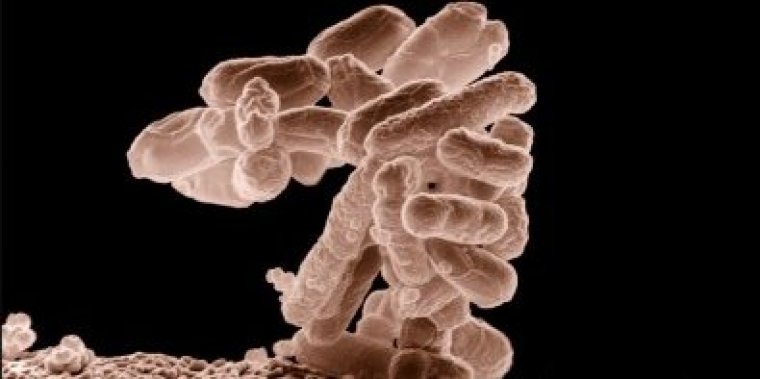
Objetivo do estudo foi avaliar o perfil de virulência e de resistência a antibióticos do principal agente de infecção do trato urinário. Foto: Ericc Erbe, Christopher Pooley/Agricultural Research Service
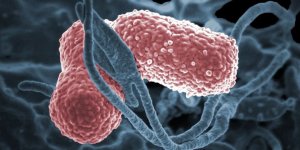
Uma das bactérias que fazem parte desse ambiente é a Escherichia coli. Praticamente todas as pessoas possuem essa bactéria no intestino, onde desempenha funções relevantes, como, por exemplo, a produção de alguns tipos de vitaminas.
“Mas existe uma diversidade genética muito grande dentro da espécie, sendo que alguns de seus membros são patogênicos, isto é, causam doenças, como, por exemplo, infecções do trato urinário”, explica Tânia Gomes do Amaral, coordenadora do Laboratório Experimental de Patogenicidade de Enterobactérias (LEPE) da Escola Paulista de Medicina da Universidade Federal de São Paulo (EPM-Unifesp).
“A E. coli é o principal agente desse tipo de infecção tanto entre indivíduos saudáveis quanto entre pessoas que estão hospitalizadas ou usufruindo de serviços associados à assistência à saúde.”
Amaral assina com 12 pesquisadores e pós-graduandos artigo sobre virulência e resistência a antibióticos dessa bactéria entre pacientes hospitalizados.
“Focamos nosso estudo na população internada, pois pacientes que permanecem hospitalizados por longos períodos têm mais chances de passar por diversos procedimentos, como, por exemplo, a utilização de sondas urinárias ou de acessos venosos. Embora esses procedimentos sejam feitos para garantir suporte à vida, podem facilitar a entrada de bactérias no corpo e promover infecção”, diz Amaral.
O trabalho publicado apresenta parte dos resultados de um estudo mais amplo, coordenado por Amaral, que avalia a virulência e resistência a antimicrobianos de cepas de E. coli associadas as infecções do trato urinário.
O principal objetivo dessa parte do estudo, que consistiu na dissertação de mestrado de José Francisco Santos Neto, foi conhecer e entender a diversidade das E. coli que colonizam o intestino de pessoas hospitalizadas, avaliando aspectos relacionados ao potencial de causar infecção e o perfil de resistência aos antibióticos.
Além disso, verificou-se quais bactérias causaram infecção nos pacientes durante o período do estudo para confirmar a frequência das chamadas infecções endógenas – causadas por bactérias da microbiota do próprio paciente.
Inicialmente, o grupo da Unifesp investigou a diversidade genética e a resistência aos antibióticos das cepas de E. coli presentes nos intestinos dos pacientes hospitalizados.
Os genomas dessas cepas e de outras isoladas da urina dos pacientes que participaram do estudo foram sequenciados. Análises comparativas foram realizadas entre as diferentes linhagens para avaliar a disseminação dos microrganismos dentro do ambiente hospitalar.
“Comparamos também os genomas dessas cepas com os de linhagens de E. coli isoladas em diferentes regiões do mundo para identificar a ocorrência, entre os pacientes do estudo, de bactérias patogênicas disseminadas globalmente”, detalha Ana Carolina de Mello Santos, pós-doutoranda do LEPE.
A equipe de cientistas conseguiu determinar a origem endógena da infecção urinária para a grande maioria dos participantes do estudo (70%). Também comprovou que os pacientes possuem, no mínimo, duas populações geneticamente diferentes de E. coli no intestino.
Outro achado foi que aproximadamente 30% das pessoas analisadas eram colonizadas por cepas de E. coli não fermentadoras de lactose (menos comuns), sendo que alguns pacientes estudados só possuíam bactérias com essa característica na microbiota intestinal.
“Esse achado é bem interessante, porque outros estudos internacionais que avaliam a composição da microbiota humana não analisam as E. coli não fermentadoras de lactose”, comenta Santos.
Outro ponto ressaltado no estudo é a presença de bactérias que contêm todos os marcadores genéticos para serem classificadas como patogênicas. Esse tipo de bactéria foi identificado no intestino de todos os pacientes que ainda não tinham desenvolvido infecção.
“Quando pensamos em pacientes hospitalizados e potencialmente mais suscetíveis a infecções devido às diversas condições de saúde que os levaram à hospitalização, a colonização por patógenos é o primeiro passo para que uma infecção possa se concretizar e impactar a estadia e manejo desse paciente no ambiente hospitalar”.
Por fim, foi identificada nos intestinos de alguns pacientes a presença de E. coli portadoras de resistência a antibióticos importantes, como cefalosporinas de terceira geração e colistina.
Em todos os pacientes em que foi verificada a colonização intestinal por bactérias resistentes a essas drogas, as mesmas bactérias também estavam causando infecção urinária endógena no paciente, ou seja, as bactérias multirresistentes estavam no intestino dos pacientes, colonizando-os, e alcançaram o trato urinário, onde causaram infecção.
“Com isso, fica evidente que uma avaliação precoce das bactérias do intestino de pacientes hospitalizados, pelo menos no caso das infecções por E. coli, pode facilitar e guiar o tratamento e identificar os pacientes que apresentam risco de evolução para doenças extra intestinais, como as infecções urinárias, que foram o foco do nosso estudo”, avalia Amaral.
“Desconhecemos se esse achado também valeria para outras bactérias que habitam a microbiota intestinal humana, como as dos gêneros Klebsiella, Enterobacter, Pseudomonas e outrasque podem causar infecções no paciente quando alcançam sítios extraintestinais.”
Em geral, bactérias desses gêneros são muito mais resistentes aos antibióticos do que as cepas de E. coli, representando um problema ainda maior para a saúde pública no ambiente hospitalar.
Outro questionamento importante levantado com o estudo está relacionado ao momento em que a colonização intestinal por essas bactérias multirresistentes e virulentas se iniciou.
Os autores do artigo não conseguiram definir se as bactérias resistentes a cefalosporinas e colistina já estavam nos intestinos dos pacientes antes da hospitalização ou se passaram a colonizar esses pacientes no ambiente hospitalar.
A análise dos genomas das cepas possibilitou a identificação de clones de “risco global” por causarem doenças graves e serem associados à resistência a antimicrobianos, conta Amaral.
"Entre esses, identificamos, nos intestinos de dois pacientes analisados, um clone idêntico a outros que foram isolados de infecções do trato urinário em Londrina, no Paraná, nos Estados Unidos e em países da Europa e Ásia. Essa identificação demonstra que algumas cepas encontradas no estudo são clones geralmente associados a infecções em todo o mundo." (Ricardo Muniz/Agência FAPESP)
VOCÊ TAMBÉM PODE ESTAR INTERESSADO EM